基因编辑进展梳理 Part II 基于CRISPR-Cas9的技术应用篇(二)
5. 优化碱基编辑器实现细胞、类器官和小鼠中的高效编辑
2018年7月,Nature biotechnology刊登了一篇新的研究,通过密码子优化和加入额外的核定位序列,重新设计BE3、BE4Gam和xBE3的序列。优化筛选的组成型和诱导型碱基编辑系统极大地提高了C to T的突变效率,重新设计的碱基编辑器还能在多种小鼠和人类细胞系以及肠道器官中进行目标修饰,该研究还在成年小鼠肝脏中成功介导了有效的体内细胞编辑。这些优化有助于单碱基编辑工具更加高效灵活的应用[13]。
6. eA3A-BE3:工程化人源APOBEC3A减少目标位点的周边突变
2018年 7月,哈佛大学和麻省总医院的J. Keith Joung课题组在Nature
biotechnology发文,通过使用一种工程化的人APOBEC3A (eA3A )结构域来减少基于CRISPR -
Cas9的胞苷脱氨酶周边突变的策略,该结构域根据一种TCR>TCY>VCN的优先等级来对特定基序中的胞苷进行去氨基化,极大地降低了普遍存在的五碱基对编辑窗口中存在不止一个C时,现有的碱基编辑器会产生不必要的C到 T转换的可能。eA3A-BE3比BE3显示更高的精度(超过40倍)以及更低的脱靶效率,且保持相似的编辑活性。利用这一系统他们还高效校正了人β-地中海贫血启动子的突变,为碱基编辑的临床运用提供了更高精度的编辑工具[14]。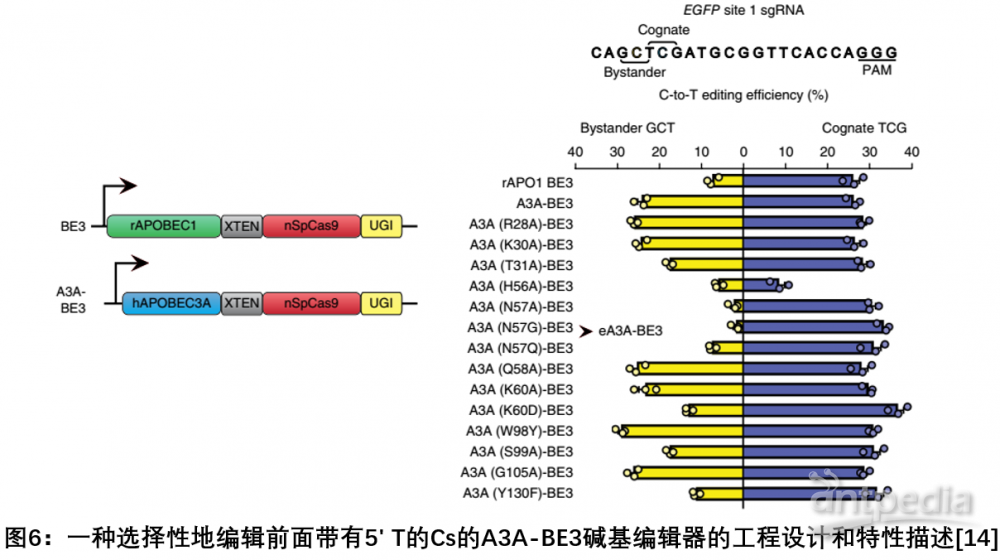
7. hA3A-BE3::人APOBEC3A实现高甲基化区域C to T的高效编辑
2018年8月,来自上海科技大学和中科院的研究人员在Nature
biotechnology发文,通过筛选多种APOBEC和 AID脱氨酶,发现人类APOBEC3A结合BEs的系统具有更窄的编辑窗口可以在甲基化水平和GpC二核苷酸含量高的区域高效介导C到T的碱基编辑,解决了目前基于大鼠APOBEC1的BEs 系统在编辑高度甲基化的胞嘧啶时效率相对较低的问题[15]。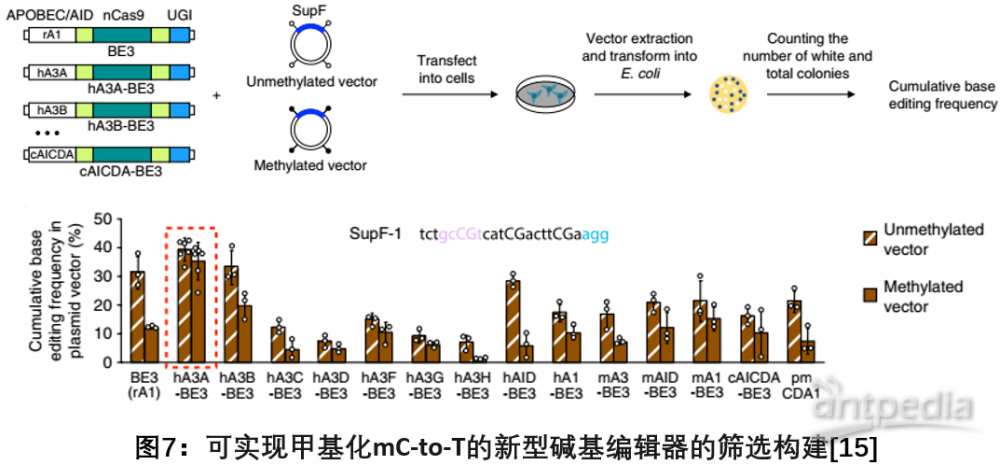
8. 同期两篇报道首次利用ABEs系统实现老鼠疾病模型的构建
2018年9月,Protein & Cell期刊刊登了两篇利用优化改良腺嘌呤碱基编辑器(ABE)分别在小鼠和大鼠中进行高效的碱基编辑。文中不仅强调了ABEs在诱导A - T转化为G - C过程中的保真度和效率,还展示了它们在产生疾病模型以及纠正动物和人类胚胎中的疾病突变方面的潜在易用性和多功能性。具体如下:
l 华东师范大学生命科学学院院长、邦耀生物首席科学家刘明耀教授及团队成员李大力教授课题组,使用优化的ABE系统有效地创建了三种小鼠品系,成功构建了遗传性肌肉变性疾病(DMD)的小鼠模型。另外同时构建了II型遗传性糖原贮积病(GSD)的大鼠模型。这是第一个通过碱基编辑系统在老鼠上实现高效点突变的报道。该研究不仅在疾病模型的构建方面有很大的潜力,更重要的是在治疗由点突变引起的遗传性疾病方面具有重要应用价值[16]。
l 中山大学黄军就和松阳洲团队同样适用ABEs系统测试了在疾病小鼠模型构建中的有效性。通过ABE来靶向mRNA剪接位点以诱导基因功能障碍的策略成功构建了小鼠白化病和DMD模型。这些结果表明ABEs可以有效和精确地将小鼠胚胎中的碱基A转化为G,证明这是一种产生点突变小鼠模型的高保真工具[17]。
9. A3A-PBE:利用nCas9和人类APOBEC3A的融合,在植物中进行高效的C to T编辑
2018年10月,中国科学院遗传与发育生物学研究所高彩霞研究组在Nature
Biotechnology上发文,展示了一种由Cas9 nickase和人APOBEC3A (A3A-PBE)组成的碱基编辑系统(A3A-PBE)在小麦、水稻和马铃薯中高效地C到T的碱基编辑。同时将尿嘧啶糖基化酶抑制剂 (UGI)和MU融合到A3A-PBE,创建了A3A-Gam系统,极大地提高了碱基编辑效率。新系统不仅将基于大鼠APOBEC1的BE3仅限5
nt序列编辑窗口扩大到了17nt,而且还能有效地编辑GC含量高的区域,对于新品种培育和作物遗传改良具有重要的应用价值[18]。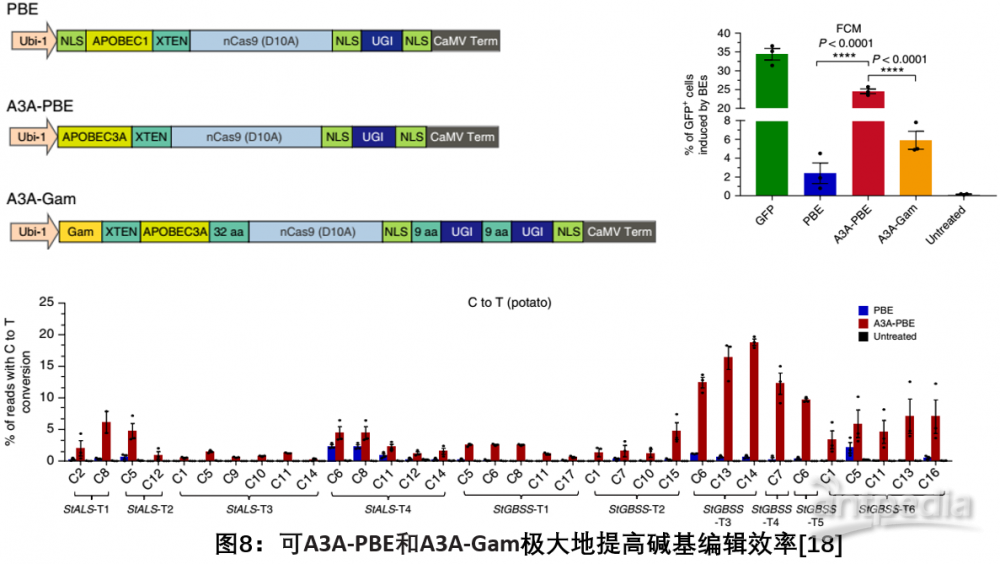
小结
综上,为大家简单介绍了2018年CRISPR系统在大动物模型构建和单碱基编辑技术上的重大突破。展望未来,我们可以预见CRISPR-Cas9技术将会给基础科研和临床研究带来的突飞猛进的发展。尽管现阶段还没有充分开发,但这项技术已经在生物以及医疗研究中带来了革命性的变革,包括基因组编辑、基因筛选和细胞示踪,疾病治疗等等。关于更多重大应用我们下期继续介绍!
参考文献:
[1] http://www.sciencemag.org/news/2017/11/human-has-been-injected-gene-editing-tools-cure-his-disabling-disease-here-s-what-you.
[2] Yan, Sen, et al. "A Huntingtin Knockin Pig Model Recapitulates Features of Selective Neurodegeneration in Huntington’s Disease." Cell (2018): S009286741830285X.
[3] Liu, Zhen, et al. "Cloning of Macaque Monkeys by Somatic Cell Nuclear Transfer." Cell (2018):S0092867418300576.
[4] Peiyuan Qiu, et al. "BMAL1 knockout macaque monkeys display reduced sleep and psychiatric disorders."National Science Review (2019): https://doi.org/10.1093/nsr/nwz002.
[5] Zhen Liu, et al. " Cloning of a gene-edited macaque monkey by somatic cell nuclear transfer." National Science Review (2019): https://doi.org/10.1093/nsr/nwz003.
[6] Yao, Xuan, et al. "Generation of knock-in cynomolgus monkey via CRISPR/Cas9 editing." Cell Research (2018).
[7] Cui, Yiqiang, et al. "Generation of a precise Oct4-hrGFP knockin
cynomolgus monkey model via CRISPR/Cas9-assisted homologous
recombination." Cell Research 28.3 (2018).
[8] Weiqi, Zhang, et al. "SIRT6 deficiency results in developmental retardation in cynomolgus monkeys." Nature (2018).
[9] Hu, Johnny H., et al. "Evolved Cas9 variants with broad PAM compatibility and high DNA specificity." Nature.
[10] Li, X. et al. Base editing with a Cpf1-cytidine deaminase fusion. Nat Biotechnol190 36, 324-327 (2018)
[11] Koblan, Luke W, et al. "Improving cytidine and adenine base
editors by expression optimization and ancestral reconstruction." Nature
Biotechnology (2018).
[12] Wen, Jiang, et al. "BE-PLUS: a new base editing tool with broadened editing window and enhanced fidelity." Cell Research (2018).
[13] Paz, Zafra Maria, et al. "Optimized base editors enable
efficient editing in cells, organoids and mice." Nature
Biotechnology (2018).
[14] Gehrke, Jason M, et al. "An APOBEC3A-Cas9 base editor with
minimized bystander and off-target activities." Nature
Biotechnology (2018).
[15] Xiao, Wang, et al. "Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion." Nature Biotechnology (2018).
[16] Yang, Lei, et al. "Increasing targeting scope of adenosine base
editors in mouse and rat embryos through fusion of TadA deaminase with
Cas9 variants." Protein & Cell 9.9 (2018).
[17] Liang, Puping, et al. "Effective and precise adenine base editing in mouse zygotes." Protein & Cell 9.9 (2018).
[18] Zong Y, et al. Efficient C-to-T base editing in plants using a fusion of nCas9 and human APOBEC3A.Nature Biotechnology (2018).




















