创新数据非依赖性采集用于复杂基质目标蛋白质...(三)
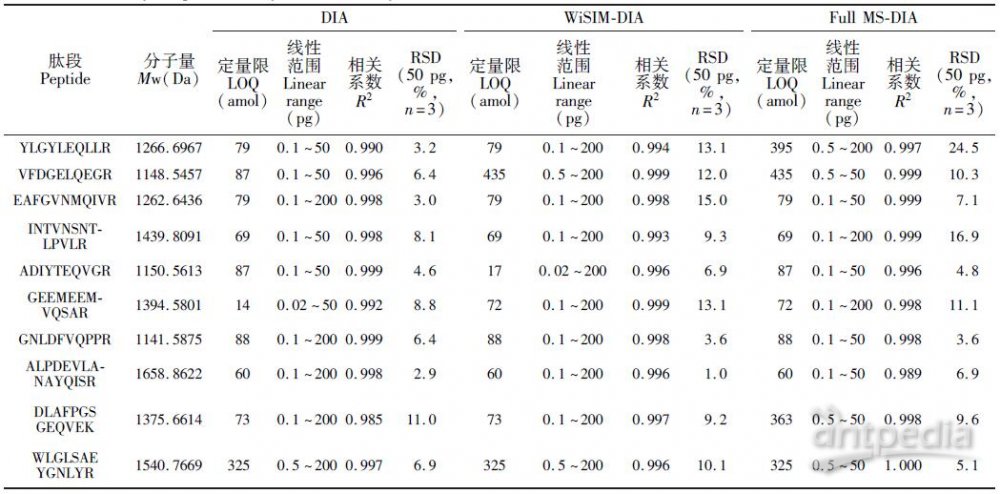
对10 条肽段的分析结果汇总(表1),结果表明,3 种DIA 方法在fg ~ pg 数量级肽段范围内均显示出良好的线性、重现性和灵敏度。3 种方法的最低定量限均达到amol 级,其中肽段GEEMEEMVQSAR在DIA 中最低定量限达14 amol,超越了常规SRM 和PRM 的定量水平。比较3 种方法可以看出,DIA与WiSIM-DIA 结果差异不大,证明了基于高分辨的二级定量和基于超高分辨的一级定量选择性相当,均能有效排除基质和共流出肽段的干扰,定量能力出色。两种方法又各有特点,形成优势互补:DIA 通过四极杆和Orbitrap 两级筛选,能有效分析极复杂的样品;WiSIM-DIA 使用母离子定量,避免了碎裂过程中的损失,在相对简单的基质中灵敏度更高。
表1 3种数据非依赖性采集的线性范围、重现性、灵敏度
Table 1 Linearity, reproducibility and sensitivity of the three DIA methods
3. 4 直接搜库鉴定的考察与比较
由于选择窗口过大,同时二级谱图无法与一级母离子相关联,传统数据非依赖性采集无法直接搜库,需要谱图库匹配才能进行定性确证,使DIA 的应用受制于DDA。
Full MS-DIA 基于一级全扫描定量,母离子未经过前级质量分析器选择,因此相比DIA 和WiSIM-DIA 受到的干扰更大,灵敏度略低。但是,Full MS-DIA 将二级选择窗口缩短到3 amu,与传统DDA 的选择窗口相当,能够作为低分辨数据,直接用于数据库检索(相当于母离子质量精度为依1. 5 amu),摆脱了谱图库的限制,实现了DDA 与DIA 的统一。
图4 展示了肽段YLGYLEQLLR 谱图的直接搜库结果。DDA 通常以2 amu 为选择窗口,与Full MS-DIA 3 amu 选择窗口相差不大。搜库时,Full MS-DIA 一级质量精度以窗口宽度为限,即依1. 5 amu,类似于低分辨质谱DDA 数据的搜库鉴定。结果显示,Full MS-DIA 与DDA 的二级谱图高度相似,虽然Full MS-DIA 谱图等同于低分辨数据,但鉴定结果没有明显差异,序列匹配完全一致。

图4肽段YLGYLEQLLR 谱图与搜库鉴定结果。(A) DDA 和(B) Full MS-DIA 的一级选择窗口和二级鉴
定图
Fig. 4Spectra and database search results of peptide YLGYLEQLLR. MS isolation window and MS/ MS identifica-tion of (A) data dependent acquisition (DDA) and (B) Full MS-DIA data
为了考察Full MS-DIA 用于搜库鉴定的可行性,实验进一步对100 ng Hela 细胞全蛋白进行DDA 和Full MS-DIA 采集,数据使用Uniprot 人类蛋白数据库搜库鉴定(表2)。其中,DDA、Full MS-DIA 一级质量精度分别为10 ppm、1. 5 Da,二级质量精度均为0. 6 Da,卡值标准为q<0. 01 (Percolator)。结果显示,DDA 共鉴定15575 条肽段,对应3228 个非冗余蛋白;Full MS-DIA 共鉴定10515 条肽段,对应2835 个非冗余蛋白。后者鉴定结果少于前者,主要是两者扫描范围不同造成的,Full MS-DIA 需考虑扫描点数,因此扫描范围较窄;此外,两者蛋白数的差异明显小于肽段数,这是由于非冗余肽段主要集中在m / z 400 ~1000 范围,扫描范围较窄对蛋白鉴定影响较小。
表2 100 ng Hela 细胞全蛋白样本DDA 与Full MS-DIA 扫描参数、搜库参数和鉴定结果(n =3)
Table 2 MS scan parameters, database search parameters and identification results of DDA and full MS-DIA analysis of 100 ng Hela cell tryptic digest (n =3)

DDA 与Full MS-DIA 的蛋白鉴定结果重合度高,表明质量精度对鉴定结果影响不大(图5A)。通过比较肽段VVIGMDVAASEFFR 的二级谱图匹配可以看出,两种方法采集的二级谱图非常相似,碎片匹配也几乎一致(图5B)。以上数据证明,Full MS-DIA 可以直接用于数据库检索鉴定蛋白,并获得可靠的鉴定结果,实现了DDA 与DIA 的统一。
图5 100 ng Hela 细胞全蛋白样本DDA 与Full MS-DIA 鉴定结果比较。(A) 两种方法蛋白鉴定结果比较;(B) 两种方法二级谱图匹配比较(肽段VVIGMDVAASEFFR)
Fig. 5 Comparison of identification results of 100 ng Hela cell digest by DDA and Full MS-DIA methods. (A) Comparison of identified number of proteins, (B) Comparison of identified MS/ MS spectra (peptide VVIGMD-VAASEFFR)

4结论
基于静电场轨道阱Q-qIT-OT 质谱建立DIA、WiSIM-DIA、Full MS-DIA 3 种数据非依赖性采集方法, 并使用添加10 条低浓度肽段的Hela 细胞全蛋白样本对方法进行考察。结果表明,3 种方法的定量限均在14 ~435 amol 范围内,线性关系与重现性良好,定性确证可靠性高。其中,WiSIM-DIA 基于超高分辨SIM 定量,与经典DIA 二级定量具有一定的互补性;而Full MS-DIA 将二级选择窗口缩短到3 amu,实现了DIA 数据直接搜库鉴定,共从100 ng Hela 细胞全蛋白样本鉴定到2835 个非冗余蛋白,与DDA 鉴定结果重合度高。基于Q-qIT-OT 的创新数据非依赖性采集方法为定量蛋白质组学提供了全新视角与策略。
References
1 Kalli A, Smith G T, Sweredoski M J, Hess S. J. Proteome Res. , 2013, 12(7): 3071-3086
2 Nagaraj N, Kulak N A, Cox J, Neuhauser N, Mayr K, Hoerning O, Vorm O, Mann M. Mol. Cell. Proteomics, 2012,11(3): M111. 013722
3 Geiger T, Wehner A, Schaab C, Cox J, Mann M. Mol. Cell. Proteomics, 2012, 11(3): M111. 014050
4 Gallien S, Duriez E, Crone C, Kellmann M, Moehring T, Domon B. Mol. Cell. Proteomics, 2012, 11(12): 1709-1723
5 Gallien S, Duriez E, Demeure K, Domon B. J. Proteomics, 2013, 81: 148-158
6 Chapman J D, Goodlett D R, Masselon C D. Mass Spectrom. Rev. , 2013: 10. 1002/ mas. 21400
7 Law K P, Lim Y P. Expert Rev. Proteomics, 2013, 10(6): 551-566
8 Gillet L C, Navarro P, Tate S, Rost H, Selevsek N, Reiter L, Bonner R, Aebersold R. Mol. Cell. Proteomics, 2012,11(6): O111. 016717
9 Egertson J D, Kuehn A, Merrihew G E, Bateman N W, MacLean B X, Ting Y S, Canterbury J D, Marsh D M, Kellmann M, Zabrouskov V, Wu C C, MacCoss M J. Nat. Methods, 2013, 10(8): 744-746
10 Senko M W, Remes P M, Canterbury J D, Mathur R, Song Q, Eliuk S M, Mullen C, Earley L, Hardman M, Blethrow J D, Bui H, Specht A, Lange O, Denisov E, Makarov A, Horning S, Zabrouskov V. Anal. Chem. , 2013, 85(24):11710-11714
Quantification Analysis of Targeted Proteins in Complex Sample by Novel Data Independent Acquisition
ZHANG Wei1 , Reiko Kiyonami2 , JIANG Zheng*1 , CHEN Wei1
1(Thermo Fisher Scientific, Shanghai 201206, China)
2(Thermo Fisher Scientific, San Jose, CA, USA)
AbstractData independent acquisition ( DIA) is a novel MS scan mode for quantitative proteomics,acquiring all precursors as well as fragments without any loss of low abundant ions, and breaks the throughput limitation of product ion quantification by high-resolution MS. Here we developed three DIA methods on quadrupole-linear ion trap-Orbitrap (Q-qIT-OT) Tribrid MS, classic DIA, as well as novel wide isolation window SIM scan (WiSIM)-DIA and full scan-DIA (Full MS-DIA). Quantitative analysis of 10 low abundant peptides spiked in Hela cell digest was performed by the three methods for linearity, reproducibility and sensitivity evaluation. The results showed that the LOQs reached amol level (14 -435 amol) with good linearity and effective MS/ MS confirmation. WiSIM-DIA utilizes ultra-high resolution SIM scan for quantification complementary with classic DIA. The isolation window of Full MS-DIA was down to 3 amu, and the data could be directly used for database searching, thus realizing the integration of data dependent acquisition (DDA) and DIA, and avoiding the limitation of using spectra library.
Keywords Orbitrap; Data independent acquisition; Proteomics; Absolute quantification
(Received 24 April 2014; accepted 4 July 2014)