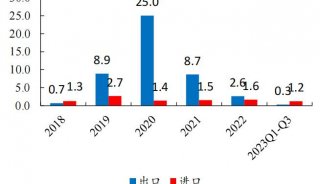
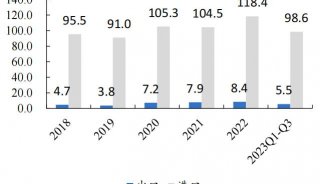
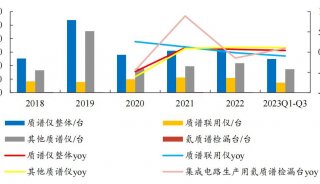
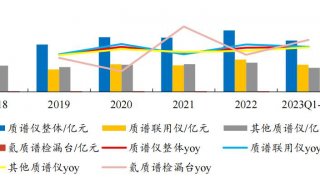
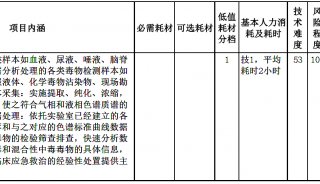
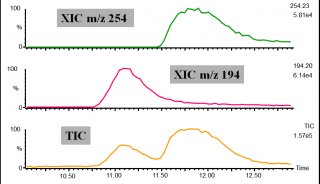
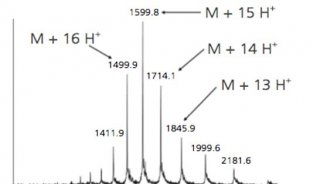
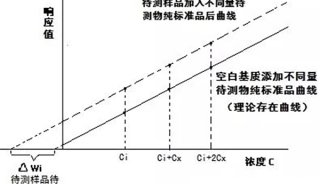
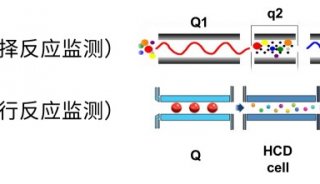
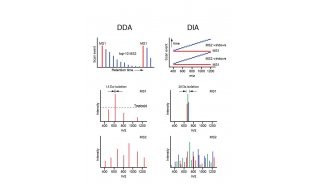
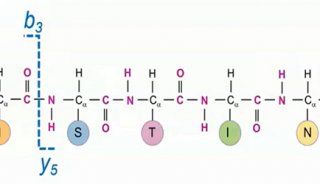
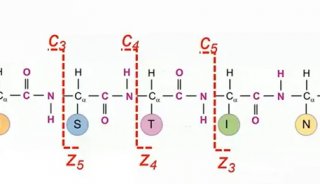
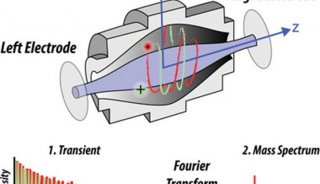
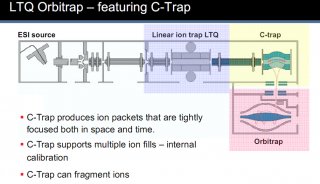
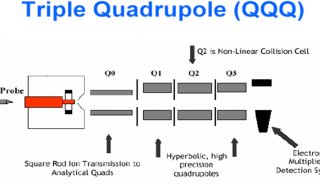
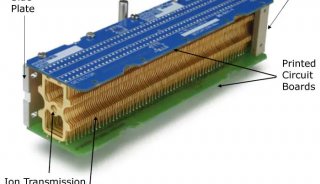
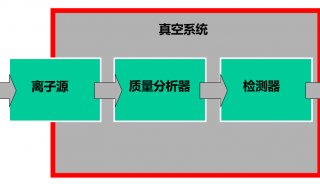
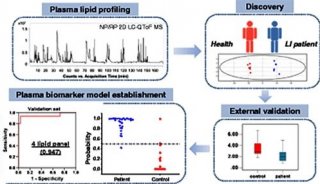
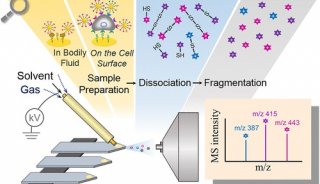
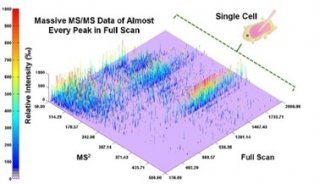
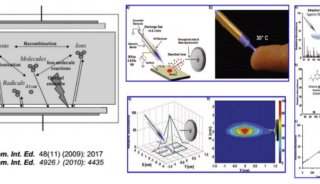
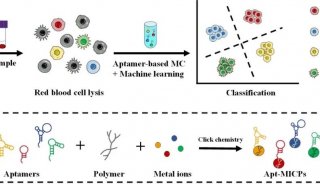
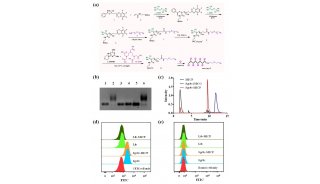
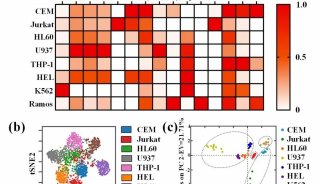
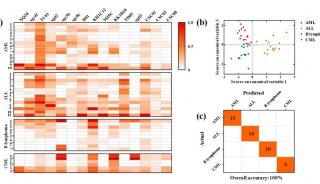
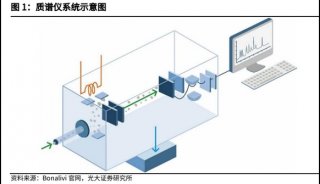
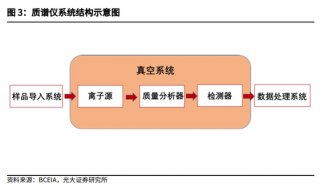
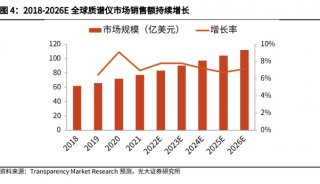
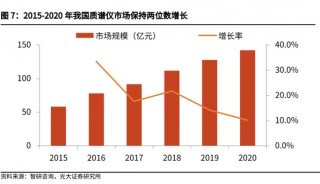
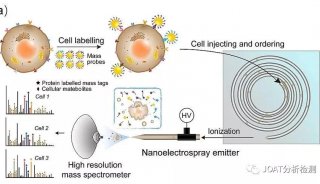
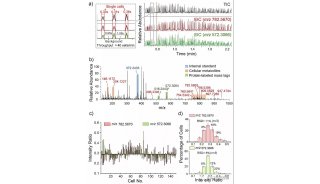
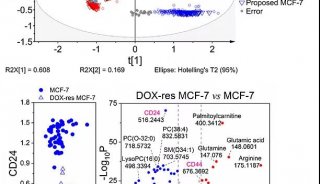
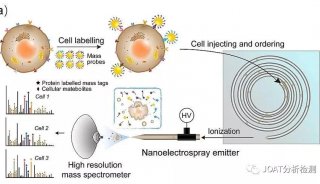
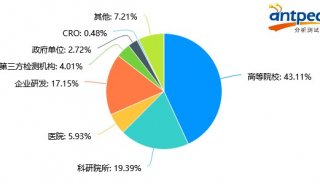
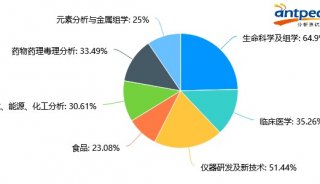
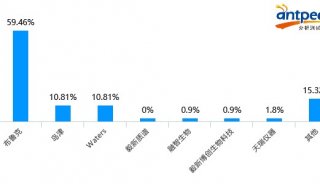
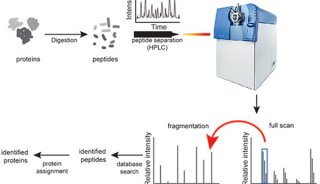
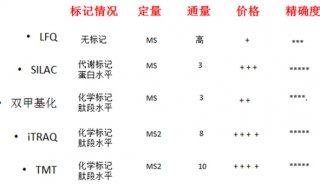
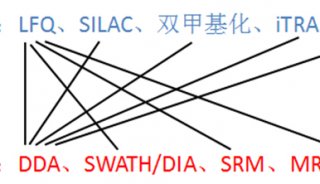
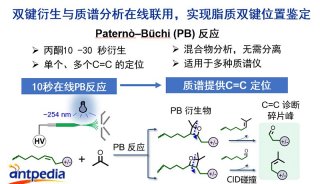
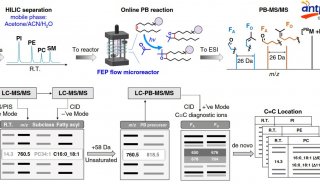
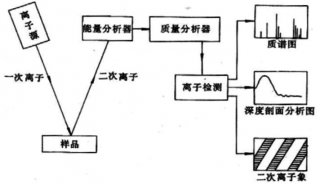
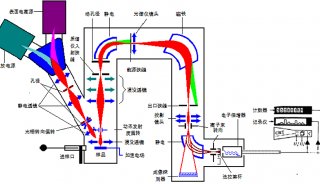
质谱分析
实验概要
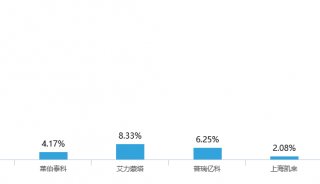
本实验在二维电泳图像获取和分析的基础上,利用质谱分析及数据库搜索鉴定部分蛋白质斑点。
实验步骤
1. 胶内酶切随机选择重复性好,差异显著,无明显变形、拖尾,与周围蛋白点分离明确的蛋白点作为质谱分析对象,进行胶内酶解。
1) 从胶上切取蛋白质凝胶颗粒置入96孔培养板内,用去离子水清洗数次。
2) 加50 ul脱色液[30 mmol/L potassium ferricyanide和100 mmol/L sodium thiosulfate 1:1]在室温下震荡至凝胶中棕黄色褪尽,弃去溶液;
3) 去离子水洗涤,重复2次;
4) 在室温下震荡10分钟脱水,吸弃其中的ACN,重复2次;
5) 加10ul/孔消化液(12.5 ng/ul胰蛋白酶和20 mmol/L NH4HCO3溶液,4℃吸胀30分钟;
6) 37oC酶解12小时以上。
2. 抽提多肽:加50ul/孔肽提取液(( 0.1% TFA和50% ACN 1:1 )室温震荡30-40分钟,收集肽提取液,置入新的96孔培养板内,真空干燥浓缩,备质谱分析用。
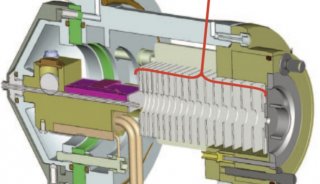
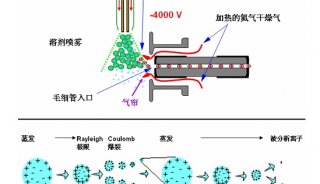
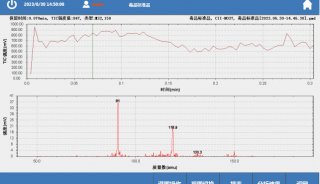
3. PMF数据采集取1ul冻干样品加1ul基质(α-CHCA饱和溶解在0.1 % TFA-30% ACN-10%丙酮溶液中)混匀后取1ul点于靶上做质谱分析。基质辅助激光解析电离飞行时间质谱( matrix assisted laser desorption/ioniz-anon time-of-flight mass spectrometry,MALDI-TOF-MS )分析采用反射模式,正离子谱测定,离子源加速电压为20 KV,每一个肽图谱由200次轰击累加形成。采用Bruker公司肽混合标准品作外校正,胰蛋白酶自降解峰(m/z 842.50;m/z 2211.10 )作内校正。用Flex Analysis 2.0 C Bruker Daltonik,Bremen,Germany)软件获得PMF。
4. 肽序列数据采集4700 Proteomics Analyzer质谱仪使用Nd:YAG激光源,激光波长355 nm,脉冲200 Hz,离子源加速电压为20KV,质量检测范围700-3,500 m/z。采用正离子模式和自动获取数据的模式采集数据。用Myoglobin酶解肽段作外标质量校正。用Data Explorer TMSoftware质谱分析软件(知plied Biosystem公司产品)处理图谱得到得到蛋白质PMF和肽序列数据。
5. 数据库查询登陆http://www.matrixscience.com,用Mascot程序对获得的PMF数据,进行数据库检索。将待测蛋白质的多个肽段质量数据与数据库中已知蛋白质肽段质量数据进行比对。如果比对的结果有显著相关性(P < 0.05 ),则蛋白质鉴定成功。检索参数如下:
Type of search Paptide Mass Fingerprint
Database NCBInr or SwissPort
Taxonomy Rattus
Enzyme Trypsin
Allow up to 1 missed cleavages
Fixed modifications Carboxymethyl(C)
Variable modifications Oxidation(M)
Max Missed Cleavages 1
Peptide tol. 土100PPM
串联质谱获得的肽段PMF和肽序列数据,通过GPS (AppliedBiosystems,USA)一MASCOT(Matrix Science,London,UK)在NCBInr数据库中搜索。检索方式为:combine。检索参数设置:种属为rattus;最大允许漏切位点为1;酶为胰蛋白酶。质量误差范围设置:PMF 0.3 Da,MS/MS 0.4 Da;手工剔除胰酶自降解峰和污染物质的峰。Mascot给出的大于95%置信水平的蛋白为鉴定成功的蛋白质。
-
招标采购

-
焦点事件

-
企业风采

-
焦点事件

-
招标采购

-
标准

-
招标采购

-
综述

-
综述
-
会议会展

-
焦点事件
-
企业风采

-
会议会展

-
焦点事件
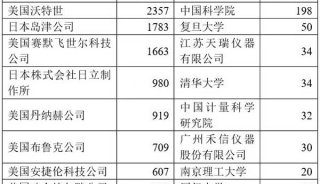
-
企业风采

-
企业风采

-
市场商机

-
精英视角

-
精英视角

-
精英视角

-
精英视角

-
精英视角

-
企业风采

-
企业风采

-
企业风采

-
企业风采

-
企业风采

-
企业风采

-
精英视角

-
会议会展

-
精英视角

-
焦点事件

-
会议会展

-
焦点事件

-
科技前沿
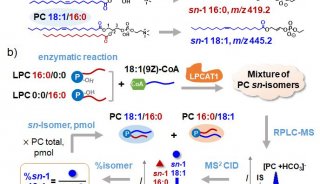
-
焦点事件
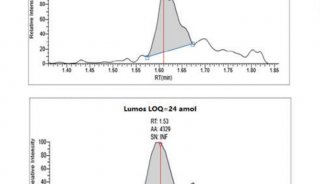
-
企业风采

-
产品技术
-
焦点事件

-
焦点事件

-
会议会展

-
企业风采

-
产品技术

-
企业风采

-
焦点事件

-
产品技术

-
焦点事件
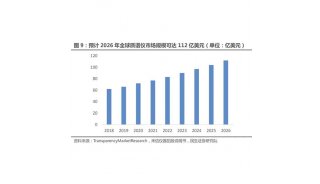
-
焦点事件

-
企业风采

-
企业风采

-
项目成果
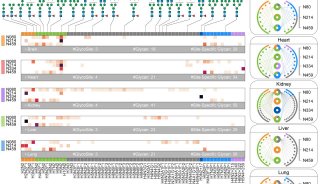
-
企业风采

-
科技前沿
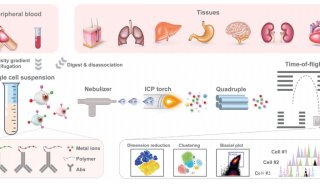
-
并购

-
会议会展

-
焦点事件

-
企业风采

-
精英视角

-
会议会展

-
企业风采

-
精英视角

-
精英视角

-
综述

-
科技前沿
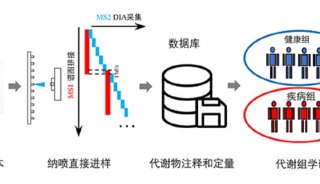
-
科技前沿
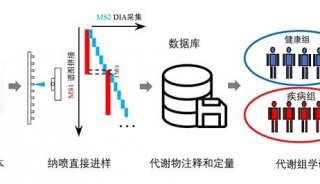
-
产品技术

-
会议会展

-
精英视角

-
产品技术

-
精英视角

-
精英视角

-
会议会展

-
项目成果
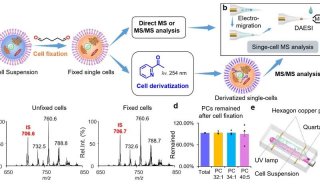
-
会议会展

-
会议会展

-
焦点事件

-
焦点事件
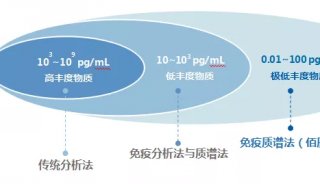
-
焦点事件

-
招标采购

-
招标采购

-
焦点事件

-
会议会展

-
会议会展

-
焦点事件

-
焦点事件

-
焦点事件

-
精英视角

-
精英视角

-
产品技术

-
焦点事件

-
综述
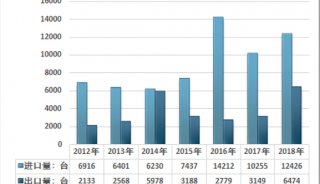
-
焦点事件

-
企业风采

-
企业风采

-
精英视角

-
会议会展

-
精英视角

-
技术原理
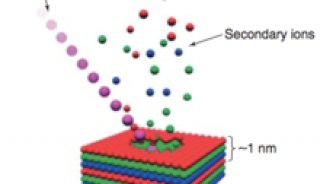
-
综述

-
焦点事件
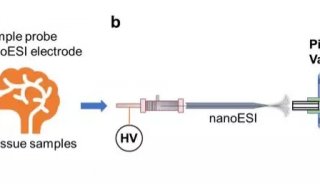
-
会议会展

-
焦点事件

-
企业风采

-
精英视角

-
焦点事件

-
精英视角

-
精英视角

-
精英视角