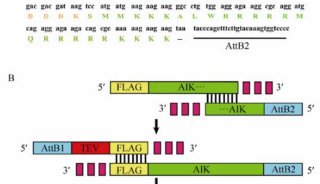
Pepfinder软件对单克隆抗体进行肽图分析(二)


图3 肽段SNWEAGNTFTCSVLHEGLHNHHTEK的脱酰胺鉴定及定量

图4 在PepFinder2.0中对所有肽段进行De Novo Sequencing

图5 对一张MS/MS谱图进行De Novo Sequencing

图6 在PepFinder软件中计算单克隆抗体各种常用分子量

图7 PepFinder软件预测的二级谱图与实测二级谱图的对比。上,预测二级谱图;下,实测二级谱图。
4结论
在本文中,我们展示了PepFinder软件对单克隆抗体进行常规肽图分析的各种应用。结果表明,使用PepFinder软件可以简便快捷的得到序列覆盖度分析的结果,并且可以对脱酰胺等生物医药质控中重要的翻译后修饰进行鉴定以及定量。PepFinder独具的单克隆抗体分子量计算功能及二级谱图智能预测功能大大减少了分析人员花费在数据处理上的时间;在PepFinder2.0版本中,新增的De Novo Sequencing功能和mgf格式文件、pinpoint txt文件输出功能帮助分析人员对数据进行不同平台上的深入挖掘。通过生成快速、全面的肽段表征及自动化的修饰位点总结报告, PepFinder 软件能够大幅减少生物产品详细表征所需的时间和精力。
参考文献
1. Z. Zhang, Automated Precursor Ion Exclusion During LC/MS/MS Data Acquisition for Optimal Ion Identification, J. Am. Soc. Mass Spectrom. 2012, 23(8), 1400-1407.
2. Z. Zhang, Large-scale Identification and Quantification of Covalent Modifications in Therapeutic Proteins. Anal. Chem. 2009, 81(20),8354-8364.
3. Z. Zhang and J. S. Mcelvain, Optimizing spectroscopic signal-to-noise ratio in analysis of data collected by a chromatographic /spectroscopic system, Anal. Chem. 1999, 71(1),39-45.
4. Z. Zhang and A. G. Marshall, A universal algorithm for fast and automated charge state deconvolution of electrospray-to-charge ratio spectra, J. Am. Soc. Mass Spectrom. 1998, 9, 225-233.
5. Z. Zhang, Retention time alignment of LC/MS data by a divide-and-conquer algorithm, J. Am.Soc. Mass Spectrom. 2012, 23(4), 764-772.
6. SC. Kim, Y. Chen, S. Mirza, et.al. A clean, more efficient method for in-solution digestion of protein mixtures without detergent or urea. J Proteome Res. 2006 Dec;5(12):3446-52.
-
焦点事件
-
会议会展

-
企业风采

-
技术原理

-
焦点事件

-
焦点事件
-
焦点事件

-
焦点事件

-
焦点事件

-
焦点事件
-
焦点事件

-
焦点事件
-
焦点事件

-
焦点事件

-
技术原理