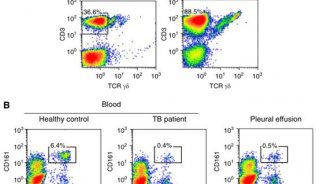
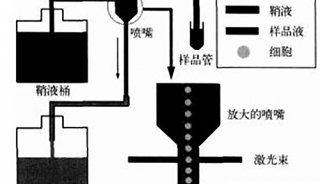
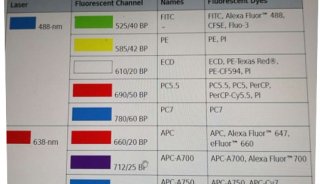
基因芯片技术的应用和发展趋势
随着基因芯片技术的日渐成熟, 在功能基因组、疾病基因组、系统生物学等领域中得到了广泛的应 用, 已经发表了上万篇研究论文, 每年发表的论文呈现增长的趋势.
芯片制备技术极大地推进了生物芯片的发展, 从实验室手工或机械点制芯片到工业化原位合成制备, 从几百个点的芯片到几百万点的高密度芯片, 生物芯片从一项科学成为一项技术, 被越来越多的研究者广泛运用. 各个实验室不断产生海量的杂交数据, 相同领域的研究者需要比较不同实验平台产生的数据, 作为基于分子杂交原理的高通量技术, 芯片实验的标准化、可信度、重现性和芯片结果是否能作为定量数据等问题成为所有的芯片使用者关心的课题. 迈阿密原则和微阵列质量控制系列研究回答了这两个问题.
迈阿密原则(Minimum Information About a Micro- array Experiment, MIAME, 微阵列实验最小信息量)提出了生物芯片标准化的概念, 该原则的制定使世界各地实验室的芯片实验数据可以为所有的研究者共 享. 同时, 美国国家生物信息学中心(NCBI)和位于 英国的欧洲生物信息学研究所(EBI)也建立了GEO (http://www.ncbi.nlm.nih.gov/geo/)和ArryExpress (http:// www.ebi.ac.uk/arrayexpress/)公共数据库, 接受和储存全球研究者根据迈阿密原则提交的生物芯片数据, 对某项研究感兴趣的研究人员可以下载到相关课题的芯片原始数据进行分析.
2006年美国FDA联合多个独立实验室进行了MAQC系列实验(micro array quality control, MAQC), 旨在研究目前所使用的芯片平台的质量控制. 该研究的12篇系列文章发表在2006年9月份的Nature Biotechnology上, 用严格的实验分析了目前主流芯片平台数据质量, 芯片数据和定量PCR结果之间的相关性, 芯片数据均一化方法, 不同芯片平台之间的可重现性. 证明了不同芯片平台产生的数据具有可比性和可重现性, 各种芯片平台之间的系统误差远远小于人为操作和生物学样品之间本身的差异, 肯定了芯片数据的可信性, 打消了以往对芯片数据的种种猜疑, 明确了基于杂交原理的芯片同样可以作为一种定量的手段. 推动了生物芯片技术在分子生物学领域更广泛的应用.
生物信息学和统计学是在处理基因芯片产生的海量数据中必不可少的工具. 随着芯片应用的推进, 芯片数据分析的新理论和新算法不断地被开发出来, 这些方法帮助生物学家从海量的数据里面快速筛选出差异表达的基因. 一次芯片实验获得的是成千上万个基因的表达信息, 任何一种单一的分析方法都很难将所有蕴含在数据中的生物学信息全部提取出来, 从近年来生物信息学研究的趋势来看, 目前研究的重点开始转向芯片数据储存、管理、共享和深度信息挖掘, 旨在从芯片数据中获得更多的生物学解释, 而不再停留在单纯的差异表达基因筛选上。
目前基因芯片的制备向两个主要方向发展. 第一, 高密度化, 具体表现为芯片密度的增加, 目前原位合成的芯片密度已经达到了每平方厘米上千万个探针. 一张芯片上足以分析一个物种的基因组信息. 第二, 微量化, 芯片检测样品的微量化, 目前芯片检测下限已经能达到纳克级总RNA水平, 这为干细胞研究中特别是IPS干细胞对单个细胞的表达谱研究提供了可能. 另一方面, 微量化也体现芯片矩阵面积的微量化, 即在同一个芯片载体上平行的进行多个矩阵的杂交, 大大减少系统和批次可能带来的差异, 同时削减实验费用.
微阵列技术改变了生物学研究的方法, 使得微量样品快速高通量的分析成为可能, 从单个基因的研究迅速扩展到全基因组的系统生物学研究. 微阵列技术帮助生物学研究进入后基因组时代, 研究成果层出不穷。
2001年国家人类基因组南方研究中心韩泽广博士研究小组利用cDNA芯片对肝癌和正常组织中的12393个基因和EST序列进行了表达谱筛查, 其中发现了2253个基因和EST在肝癌中发生了差异表达, 并对这些差异基因的信号通路进行了分析, 发现WNT信号通路在肝癌的发生中出现了表达异常. 2002年中国科学院神经科学研究所张旭博士研究组利用表达谱芯片对大鼠外周神经损伤模型背根神经节的基因表达进行了研究, 通过对7523个基因进行表达谱筛查, 发现一批与神经损伤和疼痛相关的基因和潜在的药物靶点, 并解释了临床上使用的Gabapentin治疗疼痛的分子机制. 同年Nature发表的荷兰癌症研究所的研究小组利用表达谱芯片对乳腺癌患者5年内的转移情况进行分子水平的分型工作, 研究结果显示可以利用基因表达谱的差异来预测肿瘤的预后情况. 这项工作成为用表达谱对疾病进行分子分型和预后研究的经典案例. 2006年根据这项研究成果生产的表达谱芯片成为第一张通过FDA批准进入临床使用的表达谱芯片, 迈出了芯片从实验室走向临床的第一步.
2003年启动的人类基因组单体型计划(Interna- tional Hapmap Project)采用了5个不同的高通量平台进行基因分型工作, 其中芯片完成了超过50%的工作量. 2005年Gunderson等人用DNA芯片检测人类基因组中的SNP位点, 芯片的检测结果与传统的基于PCR技术的基因分型具有非常好的相关性. Roch 的Amplichip CYP450检测芯片在2005年通过了FDA批准进入临床使用, 用以检测患者体内决定细胞色素氧化酶活性的多态性位点, 预测患者药物代谢水平的高低, 这也是第一张进入临床检查的SNP芯片. 2007年Burton 等人利用微阵列检测了1000例四种疾病患者和对照组的1500例健康人的14000多个SNP位点, 发现了患者与健康人自身免疫方面存在的基因组SNP位点差异性.
2006年国家人类基因组南方研究中心和生物芯片上海国家工程研究中心合作, 利用比较基因组杂交(aCGH)和表达谱芯片联合分析了肝癌样品中基因组拷贝数变异和基因表达量变化的相关性, 为从基因组结构变异角度研究肝癌的发生机制提供了研究基础. 2007年Carter等人报道利用SNP芯片检测基因组拷贝数变化(copy number variation), 发现人类基因组中存在约12%的拷贝数变异, 这一发现远远出乎以前人们对人类基因组多样性的预测. 2008年Lee等人报道了用比较基因组杂交(aCGH)对遗传疾病进行分型, 发现了存在于精神发育迟缓病人染色体15q13.3区域的一个缺失, Nature Genetics刊登了这一结果.
小RNA或非编码RNA(ncRNA)是新兴的一个研究领域. 这类小分子RNA在生理过程中扮演着调控分子的角色, 在发育过程和人类疾病尤其是肿瘤发生发展中起到非常重要的作用. 这些小分子核酸高度同源, 序列差异往往就是一个碱基, 且长度在21~35碱基之间, 这对芯片检测是一个很大的挑战, 要求既能检测到低分子量的目的片段, 又能区分一个碱基的差异, 芯片技术的发展已经部分解决这些问题, 利用芯片检测小分子RNA的表达谱成为肿瘤分型的另一种途径, 在乳腺癌、肺癌、前列腺癌等疾病的分子分型和分子标志物寻找中取得非常好的结果.
DNA修饰以及DNA-蛋白质相互作用是细胞对基因表达的调控方式. 2007年Meier等人采用ChIP-chip实验方法, 检测到了发生DNA损伤时, 相应的修复因子在DNA上的分布. 同年, Guenther等 人用ChIP-chip实验发现基因的转录起始可能不是特异的, 大多数的人类基因包括原来被证明转录失活的编码基因都能启动转录, 细胞通过对基因组的修饰来控制转录的延伸过程, 从而达到调控基因转录的目的. 2008年Lupien等人用ChIP-chip和一系列的实验, 证实了基因组表观遗传学修饰能调控FoxA1, 通过FoxA1改变染色质的结构, 从而控制基因的组织特异性表达.
基因芯片的应用加速了生命科学研究的进程, 微量化并行化的分析帮助科学家从海量数据中发掘有用的信息.
-
焦点事件

-
科技前沿

-
并购

-
企业风采

-
会议会展

-
会议会展

-
市场商机
-
精英视角

-
科技前沿

-
焦点事件

-
企业风采

-
焦点事件
-
焦点事件

-
焦点事件

-
焦点事件

-
焦点事件

-
技术原理