Science发表单分子成像新应用 年前将入职西湖大学
真核生物基因组极具复杂性。如何将宏观尺寸的基因组DNA包装入微米级的细胞核中是生命科学的基本问题之一。在细胞周期中,基因组具有高度组织性。在细胞周期间期,基因组形成不同的区室(Compartment)和结构域(Domain),而在有丝分裂期,基因组被高度凝聚成X形染色体。一类高度保守的ATP酶多亚基蛋白质复合物——SMC复合物,在基因组的三维结构中扮演中关键的角色【1-3】。真核生物的SMC复合物包括粘连蛋白(Cohesin),凝缩蛋白(Condensin)以及SMC5/6复合物。粘连蛋白的主要功能是介导间期染色质环结构域(Loop domain)和拓扑相关结构域(Topologically associating domain,TAD)的形成以及细胞分裂中姐妹染色单体的粘连。凝缩蛋白介导有丝分裂中的染色体凝聚。粘连蛋白和凝缩蛋白一起保证了有丝分裂末期姐妹染色单体的正常分离。虽然SMC复合物对细胞活动至关重要,然而其作用机制仍不清楚。
人类粘连蛋白由SMC1,SMC3,RAD21以及STAG1/2四个亚基组成,并且能够形成环状结构。在装载蛋白NIPBL-MAU2的帮助下,粘连蛋白加载到染色体上发挥功能【4,5】。NIPBL的突变会引起发育相关疾病包括德朗热综合征(Cornelia de Lange syndrome)的发生。大量证据表明粘连蛋白与CTCF一起调控有丝分裂间期基因组组织和基因转录,并提出粘连蛋白通过环挤压(Loop extrusion)方式来介导染色质结构域的形成【6,7】。2018年初,研究人员在体外观测到酵母凝缩蛋白介导的DNA环挤压过程【8】。然而对于粘连蛋白是否具有该活性,仍缺乏直接的证据。
2019年11月29日,来自美国得克萨斯大学西南医学中心于洪涛教授课题组与得克萨斯大学奥斯汀分校的Ilya J. Finkelstein教授课题组合作,在Science上在线发表题为Human cohesin compacts DNA by loop extrusion的研究论文(Yoori Kim博士和史竹兵博士为共同第一作者,Hongshan Zhang博士为共同作者),他们利用单分子成像技术在体外实时观测到重组的人源粘连蛋白-NIPBL复合物可以压缩裸露的以及核小体结合的DNA,并证明粘连蛋白-NIPBL复合物通过ATP驱动的环挤压方式来压缩DNA。值得一提的是,来自奥地利的Jan-Michael Peters课题组近期也有类似的发现【9】。另据悉,于洪涛教授会于2019年12月份全职回国工作,在西湖大学组建实验室。

在于洪涛课题组的研究中,研究人员首先克服了人源粘连蛋白以及粘连蛋白-NIPBL复合物表达与纯化的困难,获得高质量的蛋白样品用于单分子成像研究。体外酶活实验发现,粘连蛋白本身具有非常低的ATP酶活性,而NIPBL和DNA可以显著提高其ATP酶活性。负染电镜结果显示粘连蛋白-NIPBL复合物具有多种构象,可能代表ATP水解以及DNA结合时的不同状态。
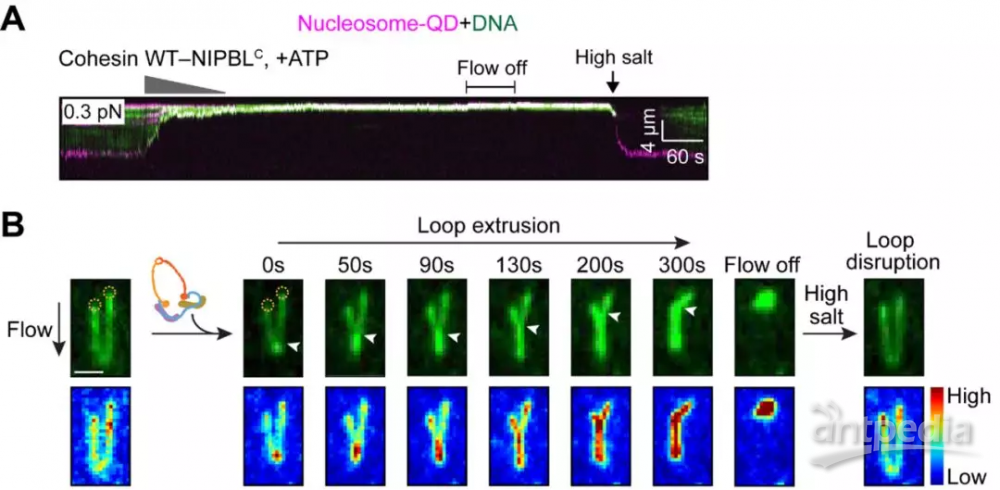
令人振奋的是,研究人员利用全内反射荧光显微镜,实时观测到粘连蛋白-NIPBL复合物可以作为分子马达压缩固定于脂双层表面的线形DNA。在未加粘连蛋白-NIPBL复合物时,这些线性DNA通过溶液流动得到完全伸展。粘连蛋白-NIPBL复合物非常有效地以0.5 kb/s的平均速率压缩DNA,并且这一过程依赖于ATP水解,而单独的粘连蛋白不具有DNA压缩功能。通过改变溶液流速进而改变DNA所受应力,研究人员发现应力的增加导致DNA压缩速率的降低,说明粘连蛋白-NIPBL复合物介导的DNA压缩是力敏感的。更为重要的是,人们一直认为粘连蛋白通过拓扑结合方式将DNA捕获在环状结构里来发挥功能,然而研究人员发现高浓度盐缓冲液可以逆转DNA压缩过程,暗示粘连蛋白是以非拓扑DNA结合方式来压缩DNA的。为了模拟体内的染色质状态,研究人员在上述实验的基础上引入核小体。结果显示,与裸露DNA相似,粘连蛋白-NIPBL复合物以相同的速率压缩核小体结合的DNA。这一过程同样依赖于ATP水解,而且也是力敏感的。这些结果表明,在细胞内,核小体的存在不影响粘连蛋白引发的染色质压缩(图一)。
图一
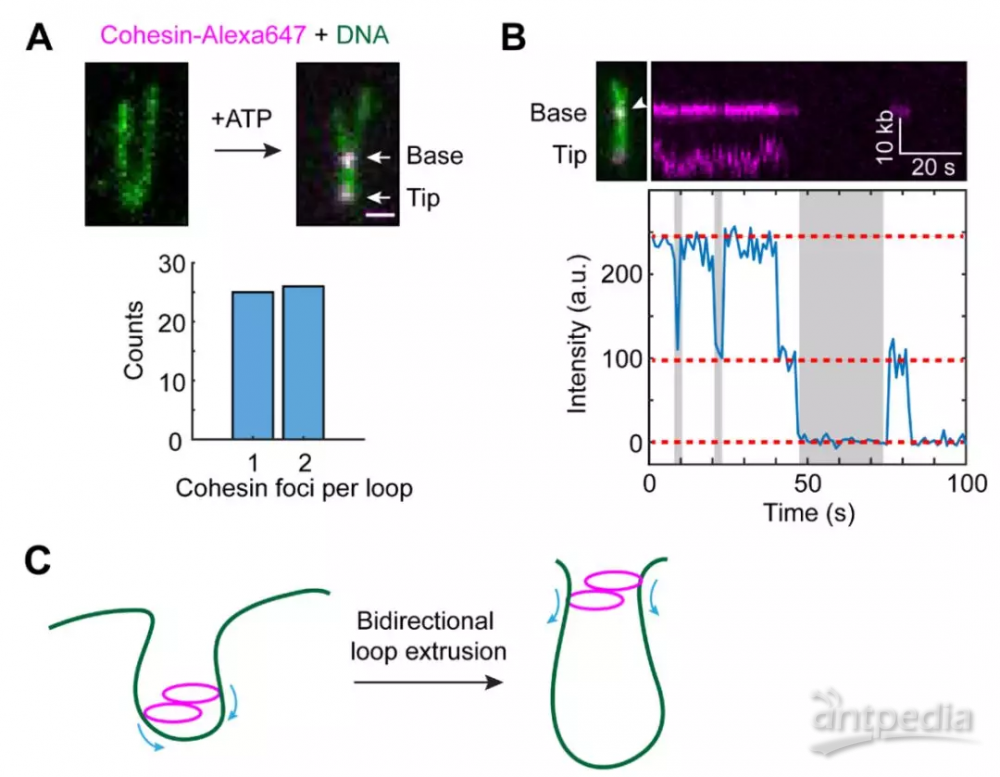
进一步地,研究人员制备了两端锚定的“U”型DNA分子来直接观察DNA环挤压过程。当将粘连蛋白-NIPBL复合物注入“U”型DNA中,DNA环开始从其顶部开始伸出,直至DNA的一侧锚定点(图一)。值得注意的是,之前的研究显示凝缩蛋白以不对称的方式来进行DNA压缩,然而于洪涛课题组的研究发现粘连蛋白-NIPBL复合物以对称的方式从“U”型DNA两侧同时压缩DNA。粘连蛋白-NIPBL复合物介导的DNA环挤压过程以0.5 kb/s的平均速率持续作用到数万碱基对上,并且最大速率可达到1.3 kb/s。通过荧光标记粘连蛋白并利用光漂白实验发现,粘连蛋白-NIPBL复合物与DNA环基部共定位,并在多数情况下粘连蛋白-NIPBL复合物以二聚体的形式发挥功能(图二)。这与已报道的凝缩蛋白亦不相同。凝缩蛋白以单体的形式单方向不对称地压缩DNA,而粘连蛋白以二聚体的形式双向对称地介导DNA环挤压过程。因此,虽然SMC复合物具有相似的结构,然而具体的作用机制截然不同。在原核生物中,仅有一种SMC复合物存在。体内的证据显示原核SMC复合物也可能是以二聚体形式发挥DNA压缩功能。因此,在进化过程中,生命体生成不同的SMC复合物,它们在特定的时期以不同的方式行使相似但特异的生物学功能。
图二
综上,这项研究为粘连蛋白环挤压模型提供了直接的实验证据。粘连蛋白-NIPBL复合物作为ATP水解驱动的分子机器,以二聚体作为基本功能单元,非拓扑形式结合并双向压缩DNA,形成对称的DNA环结构域。这一结果与已报道的高通量染色体构象捕获技术(Hi-C)数据,细胞内相关研究以及理论模拟模型相吻合。考虑到粘连蛋白的重要生物学功能,这项研究为进一步深入理解真核基因组结构和基因转录调控机制奠定了基础,也为相关疾病发生机理的阐明提供了理论依据。
原文链接
https://science.sciencemag.org/content/early/2019/11/25/science.aaz4475
制版人:小娴子
参考文献
1. Jeppsson, K., et al., The maintenance of chromosome structure:positioning and functioning of SMC complexes. Nat Rev Mol Cell Biol, 2014. 15(9): p. 601-14.
2. Rowley, M.J. and V.G. Corces, Organizational principles of 3D genomearchitecture. Nat Rev Genet, 2018. 19(12):p. 789-800.
3. Uhlmann, F., SMC complexes: from DNA to chromosomes. Nat Rev Mol Cell Biol,2016. 17(7): p. 399-412.
4. Ouyang, Z. and H. Yu, Releasing the cohesin ring: A rigid scaffoldmodel for opening the DNA exit gate by Pds5 and Wapl. Bioessays, 2017. 39(4).
5. Zheng, G. and H. Yu, Regulation of sister chromatid cohesionduring the mitotic cell cycle. Sci China Life Sci, 2015. 58(11): p. 1089-98.
6. Nasmyth, K. and C.H. Haering, Cohesin: its roles and mechanisms. AnnuRev Genet, 2009. 43: p. 525-58.
7. Alipour, E. and J.F. Marko, Self-organization of domain structures byDNA-loop-extruding enzymes. Nucleic Acids Res, 2012. 40(22): p. 11202-12.
8. Ganji, M., et al., Real-time imaging of DNA loop extrusion bycondensin. Science, 2018. 360(6384):p. 102-105.
9. Davidson, I.F., et al., DNA loop extrusion by human cohesin.Science, 2019.
-
科技前沿

-
科技前沿

-
科技前沿

-
焦点事件

-
焦点事件

-
焦点事件

-
技术原理