Nat Microbiol:新研究表明可对微生物群落进行CRISPR/Cas编辑
迄今为止,CRISPR酶已被用于一次编辑一种细胞类型的基因组:它们对一种组织或器官内的特定细胞类型或者在试管中生长的一种微生物进行基因切割、剔除或添加。
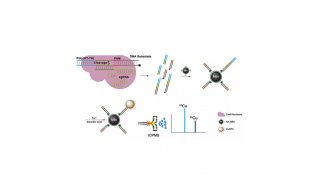
如今,在一项新的研究中,来自美国加州大学伯克利分校的研究人员找到了一种方法,可以同时在许多不同物种的群体中添加或修改基因,这就为所谓的“群落编辑(community editing)”打开了大门。相关研究结果于2021年12月6日在线发表在Nature Microbiology期刊上,论文标题为“Species -and site-specific genome editing in complex bacterial communities”。
虽然这项技术仍然只在实验室环境中应用,但是它可以用来编辑自然群落---聚集了数百或数千种不同微生物的肠道或植物根部---中的微生物,并对自然群落中经过编辑的微生物进行追踪。当科学家们谈论从基因上改变微生物种群时,这种追踪变得很有必要:例如,将基因插入肠道中的微生物以解决消化问题,或者改变农作物的微生物环境以使其对害虫有更强的抵抗力。
由于没有使用条形码来追踪基因插入的方法,这些插入的基因可能会出现在任何地方,因为微生物之间经常分享基因。
论文共同第一作者、加州大学伯克利分校博士后研究员Benjamin Rubin说,“破坏和改变分离的微生物中的DNA对于理解它们的DNA的作用至关重要。这项研究有助于将这种基础方法带入微生物群落,毕竟微生物群落更能代表这些微生物在自然界的生活和功能。”
虽然同时能够“猎枪(shotgun)”编辑多种类型的细胞或微生物的能力在目前的工业规模系统中可能是有用的,例如用于培养大量细胞的生物反应器,但更直接的应用可能是作为了解细菌、古细菌和真菌的复杂群落结构以及在这些不同微生物种群中的基因流动的工具。
论文共同第一作者、加州大学伯克利分校博士后研究员Brady Cress说,“最终,我们可能能够消除你的肠道细菌中导致疾病的基因,或者通过对它们的微生物伙伴进行基因改造使植物更有效率。但是很可能,在我们这样做之前,这种方法将使我们更好地了解微生物如何在一个群落中发挥作用。”
从普查到编辑
论文共同第一作者、加州大学伯克利分校创新基因组学研究所项目科学家Spencer Diamond在Jill Banfield实验室从事研究工作。Banfield开创了群落测序或者说宏基因组学领域:对复杂的微生物群落中的所有DNA进行鸟枪法测序,并将这些DNA组装成所有这些微生物的完整基因组,其中的一些微生物可能以前从未见过,而且许多微生物不可能在实验室的培养皿里生长。
在过去的15年里,宏基因组测序取得了巨大的进步。2019年,Diamond从加州北部的一个草原草甸上收集的土壤样本中组装了近800种微生物物种的10000个基因组。
但他把这比作进行人口普查:它提供了无可比拟的信息,说明哪些微生物以何种比例存在,以及这些微生物在群落内可以发挥哪些功能。它允许你推断这些微生物之间复杂的相互作用,以及它们如何一起工作以实现重要的生态系统效益,如固氮。Diamond说,这些观察只是假设;需要新的方法在群落水平上实际测试这些功能和相互作用。
他说,“有这样一个关于代谢传递(metabolic handoffs)的想法---没有任何一个微生物在执行一连串巨大的代谢功能,但在大多数情况下,每个单独的微生物在执行一个过程的单一步骤,而且微生物之间必须有一些代谢物的传递。这是假设,但我们如何实际证明这一点?我们怎样可以进行一些操作,看看发生了什么?这就是群落编辑的起源。”
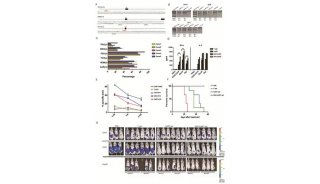
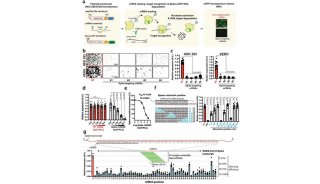
在这项新的研究中,这些作者首先开发了一种方法,以确定一种群体中哪些微生物实际上容易受到基因编辑的影响。Rubin和Diamond开发的筛选技术,即ET-seq(环境转化测序),使用容易随机插入到许多微生物基因组中的转座子(一种跳跃基因)作为探针。通过对引入转座子之前和之后的群落DNA进行测序,他们能够确定哪些微生物能够整入转座子基因。该方法是基于论文共同作者Adam Deutschbauer在劳伦斯伯克利国家实验室开发的技术。在一项涉及由九种不同微生物组成的微生物群落的实验中,他们使用不同的转化方法成功地将这种相同的转座子插入到其中的五种微生物中。
Cress随后开发出一种称为DART(DNA-editing All-in-one RNA-guided CRISPR Cas Transposase, DNA编辑一体式RNA引导的CRISPR Cas转座子酶)的靶向递送系统,它使用一种类似于CRISPR-Cas9的CRISPR-Cas酶来靶向特定的DNA序列,并插入经过条形码标记的转座子。
为了用更真实的微生物群落来测试DART技术,这些作者从婴儿身上提取的粪便样本并将其培养成一种主要由14种不同类型的微生物组成的稳定群落。他们能够在这种群落中对大肠杆菌菌株进行编辑,以便靶向与疾病有关的基因。
这些作者希望利用这一技术在一种封闭系统中了解人工的、简单的群落,如植物及其相关的微生物群。他们随后就可以在这种封闭系统中操纵群落中微生物的基因,并跟踪对它们的经过条形码标记的微生物的影响。
-
科技前沿

-
项目成果

-
焦点事件

-
焦点事件

-
焦点事件

-
科技前沿

-
科技前沿

-
市场商机

-
焦点事件

-
焦点事件
-
项目成果

-
焦点事件

-
焦点事件

-
焦点事件

-
科技前沿
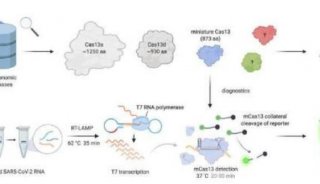
-
科技前沿

-
科技前沿

-
科技前沿

-
项目成果
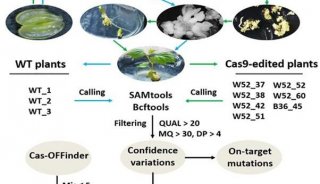
-
项目成果
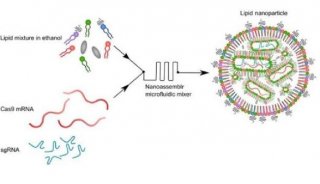
-
焦点事件

-
项目成果
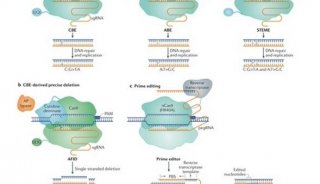
-
科技前沿
-
项目成果
-
科技前沿
-
科技前沿
-
焦点事件

-
焦点事件

-
焦点事件

-
科技前沿
-
项目成果
-
焦点事件
-
项目成果
-
焦点事件
-
项目成果
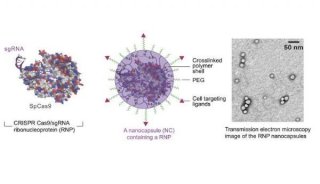
-
项目成果
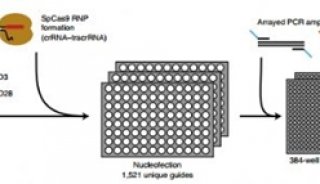
-
科技前沿

-
项目成果
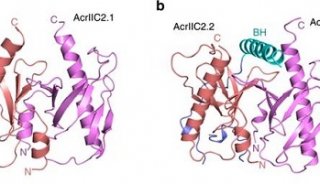
-
焦点事件
-
项目成果
-
项目成果
-
科技前沿
-
项目成果
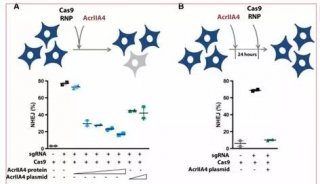
-
并购

-
科技前沿
-
焦点事件

-
焦点事件

-
技术原理

-
技术原理
-
技术原理
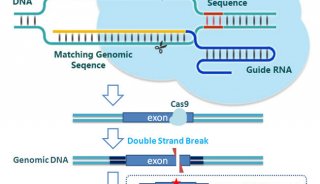
-
政策法规

-
项目成果
-
焦点事件

-
焦点事件
-
精英视角

-
焦点事件
-
焦点事件

-
焦点事件
-
焦点事件
-
焦点事件
-
科技前沿
-
科技前沿
-
项目成果
-
焦点事件
-
科技前沿

-
焦点事件

-
科技前沿
-
项目成果
-
项目成果
-
项目成果