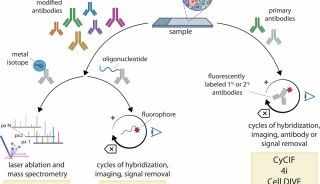
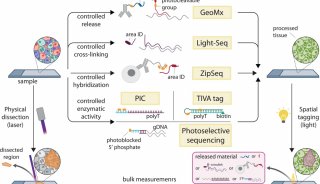
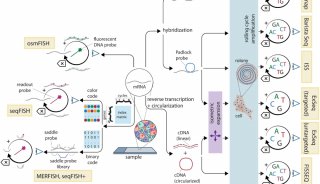
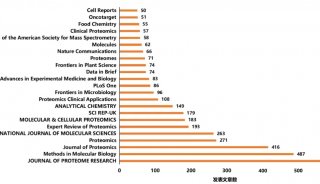
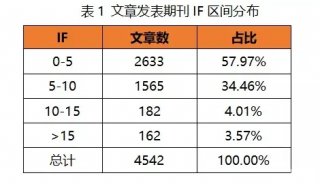
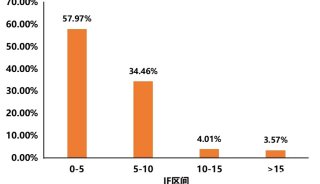
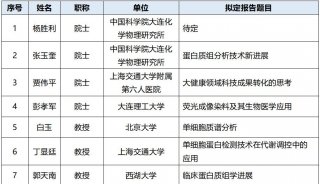
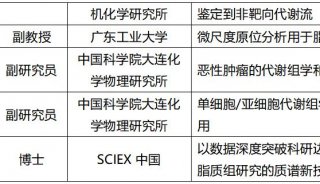
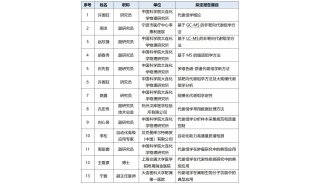
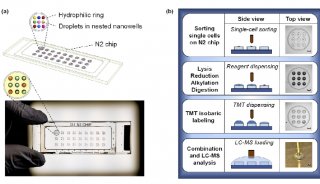
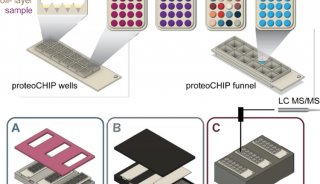
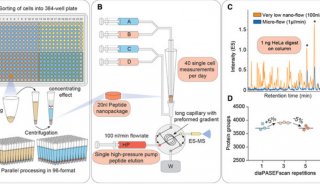
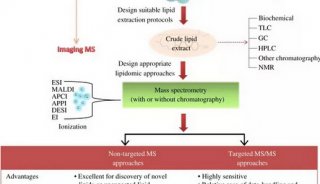
实质等同性(转录组学)实验2
3.9 芯片数据介绍
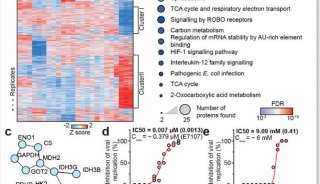
对简单的实质等同性实验来说,一个比较转基因系与对照之间基因表达的散点图就已足够了。文献 [ 5 ] 中参与两个实验的样品都标注在图15. 2中。结果用 GeneSpring 软件包显示,绘制了每组比较小麦系之间每个基因成对的平均强度,并突出显示少数感兴趣的基因(统计上显著差异表达)。结果也在表15 . 2 中作了数值化总结。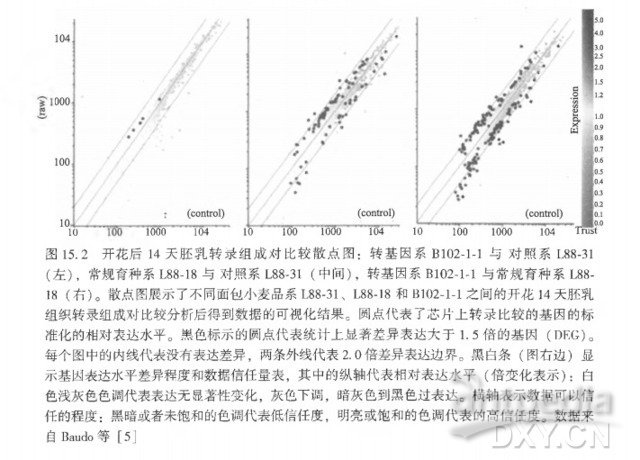
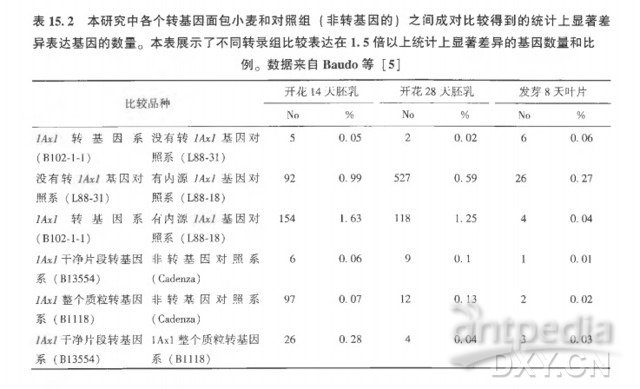
结果表明,转基因并未影响显著数量的内源性基因的表达,转基因植物实质等同于其相应的非转基因对照或亲本[ 5 ] 。结果也证实了转化方法( 如干净片段或者整个质粒)对基因表达模式影响不大。实质等同性实验强调统计上的严谨性,对 cDNA 芯片来说 ,考虑诸如染料偏好性和芯片空间变异等的问题( 见注20,目前 cDNA 芯片实验数据分析的评价)。
对于简单的实质等同性实验而言,一张简单的比较转基因系和对照系之间表达的散点图就可以了,更复杂的设计可能需要其他的显示方式。无论是 cDNA、寡核苷酸芯片或者其他平台,分层聚类是一种有力的转录组数据概览方法 [16]。这种方法将有基因关联表达的样品和(或)基因分组,不同的关联程度以树状图可视化。它通常是以热图样式显示的:基因树在一边,样品树在另一边,颜色代表基因的表达量。如果检测到处理对表达有影响,不同处理的样品会表现为不同的分支,所有重复出现在这些分支内的叶片上;在另一维,不同表达的基因也会聚在一起。
基因表达谱的非分层聚类方法,如 K 均值、质量阈值(QT ) 和自组织映射等也是常用到的。基因簇的平均表达值可以简化转录组数据。共表达意味着共同的转录调控和潜在的功能关系。进一步检查基因簇内基因,以确定是否有任何共同的已知功能(如蛋白质储存、应激反应或防御),或参与共同途径。对于作物如小麦中的绝大多数基因,功能只可能从序列相似性推测。聚类分析,显示和注释工具可以在开放资源 [ 如 Bioconductor (http://www .bioconductor .org / ) ] 和商业软件包 [ 如 GeneSpring (AgilentTechnologies, Inc) ] 获得。
3.10 分析小麦基因芯片背景
这里概述了 Affymetrix 基因芯片表达分析应遵循的步骤。详细的标准流程可以在“ Affymetrix 基因表达分析技术手册”中查询(见注 21 ) 。
基因芯片探针芯片是由 Affymetrix 公司制造的(见注 22) 。现在许多大学和私入公司已全面配备 Affymetrix Gene Chip TM 芯片平台,为客户提供各种芯片检测过程服务(GeneChip芯片探针购买、cDNA 标记、芯片杂交、扫描、芯片分析等)。
Affymetrix Wheat Gene Chip 芯片由 AffymetrixGene Chip Consortia Program 制造,包含了 61127 套探针,代表小麦基因组所有 42 条染色体上 55052 个转录本。芯片是基于 GenBank 和 dbEST ( http://www.affymetrix.com/community/research/consortia .affx ) 上发表的功能域数据设计的。小麦基因组芯片可用于不同小麦种的基因表达研究: UniGene Build 38,2004 . 4 . 24 ) 。该芯片包含了至 2004 年 5 月,所有这些种的 EST 和全长序列设计的探针。
GeneChip 探针芯片制造过程结合了光刻和化学合成技术。1.7cm2 的芯片上有几万到几十万个不同的寡核苷酸探针,每个探针点(探针室)20 mm。每一个目标转录组由一套长度 25 个碱基的 11 个 PM 和 11 个 MM 探针来检测。这些 PM 和 MM 探针(探针对)位置彼此相邻。基因表达水平可以用 Affymetrix 软件通过 PM 和 MM 探针之间亮度差异来计算(见注22) ,或者只用 PM 探针的亮度计算(RMA 和 gcRMA 分析)。
3.11 小麦基因芯片表达分析
3.11.1 RNA 样品准备
对于特定组织,RNA [ 总 RNA 或者提纯的 poly ( RNA species ) ] 提取和纯化可以采用已有的步骤(步骤与 3. 4节叙述的类似)。RNA 提取也有许多商业试剂盒可供选择。例如,TRIZOL - Reagent (Invitrogen ,见注2 3 ) 被推荐为小麦旗叶的总 RNA 提取方法。当提取物含有大量的糖蛋白和多糖时,标准步骤中匀浆(TRIZOLRNA提取说明步骤1 ) 和 RNA 沉淀(TRIZOLRNA 提取说明步骤 3 ) 需要略作修改。匀浆这一步,匀浆产物需要增加一步离心(见注 24) 。RNA 沉淀这一步,水相回收沉淀总 RNA 需要用异丙醇和高盐沉淀溶液(见注 25) 。为了得到高纯度 RNA ( 特别是 A260/A230。比值 >1.8),我们还建议 RNA 过 RNeasy 柱(Qiagen,见注26 ) 洗涤。RNA 清理建议放在总 RNA 样品中去除基因组 DNA 之后进行(见 3.4 节)。核酸浓度和质量分别用 Nanodrop ND 1000 分光光度计和 Aglient 2100 生物分析仪(RNA 6000 Nano Assay, Agilent Technologies , Palo,Alto,CA , USA ) 检测(见注 16)。
3.11.2 cDNA 合成和标记(见注 27 )
从总 RNA [ 或者纯化的 poly ( A ) RNA ] 合成双链 cDNA,然后生物素标记的 cRNA 由 cDNA 体外转录。与基因探针芯片杂交前,发现 cRNA 片段对于最高灵敏度是很关键的。
3.11.3 杂交(见注 28)
准备杂交混合物包括 cRNA 片段和探针芯片对照。与探针芯片杂交,孵育 16 h。
3.11.4 探针芯片洗脱与染色
( 1 ) 流体工作站(fluidic station ) 的设置: 流体工作站是用于芯片洗脱和染色的。它是通过兼容 PC 工作站上 GeneChip Operating System ( GCOS ) /Microarray Suite 操作的。步骤包括设置和启动流体工作站(见注 29 和注 30)。
( 2 ) 芯片的洗涤和染色(见注 31 ) : 16 h 杂交后,除去芯片上杂交液( 见注 28) ,然后将芯片完全浸入适当体积推荐的清洗液(见注 31)。
3.11.5 芯片扫描(见注 31)
一旦扫描结束,每张完整的芯片图保存在一个以.dat 为扩展名、实验名字命名的文件。GCOS 采集和分析芯片图谱及实验数据:定义探针单元并计算每个单元的光强度 ( 见注 22) 。由于制造过程中的更高的质量控制,许多 cDNA 芯片图谱分析的问题,如空间变异对 Affymerix 芯片来说不用考虑( 3.8 节)。产生了包含每一个 PM 和 MM 探针信号值的输出文件(cel 文件)。
3.12 小麦基因芯片数据分析
Affymetrix 公司芯片广泛用于许多种生物,人们投入了相当大的精力来开发和测试不同的方法分析信号,以寻找最好的基因表达检测方法。Affymetrix 开发的方法是以一组探针的 PM 和 MM 之间的平均差异来估计表达( MAS5 ) 。然而,MM 信号值的有效性还有疑问,而且有一些替代的方法看起来事实上超过了 MAS5。RMA (robustmultichipaverage) 算法取一个实验的所有 PM 数据(如全部 cel 文件),不仅标准化每个芯片表达数据中位数,而且对每个芯片表达数据使用相同的方差 [17]。gcRNA 算法是 RMA 一个变种,它将每个探针的 GC 组成对信号贡献的权重考虑在内[18]。与其他方法相比,在标样 [ 19 ] 检测和实时反转录 PCR 定量 [ 20 ] 方面,它能很好处理 Affymetrix 芯片数据。gcRNA 算法可以使用开源的 Bioconductor 软件包(http :// w w w .bioconductor .org) 或者商业软件如 GeneSpring7 (Agilent Technologies, Inc) 。
一旦选择了表达检测方法(如 MAS5、RMA 、gcRNA 或者其他),接下来的分析是一样的,而非标准化数据( 如 MAS5 ) 必须首先进行标准化(如除以每一个芯片表达值的中位数)。建议先筛选探针集,保留绝对表达值高于阈值(至少一个样品如此)的所有探针(可以从芯片中非小麦对照的信号判断)。这些探针进一步筛选那些表达差异在任何一对样品的阈值之上的;通常 1.4 倍的变化被认为是可以检测到的最小值。实质等同性实验设计通常有 2 个以上基因型,至少 3 次生物学重复。为了检测到基因型之间统计上显著差异表达的基因,对每个探针集的表达值的对数(它们通常呈对数正态分布)作方差分析 (ANOVA ) 。假定即使经过筛选,探针数量依然非常大,可以用多重检验校正。Benjamini-Hochberg 假阳性率( FDR ) 校正 [ 21 ] 是不错的选择。对许多探针做经典的 P<0.05 的方差分析和 Benjamini-Hochberg 多重检验校正,能够有大约 5% 的基因通过纯属偶然。然而,如果很少或根本没有基因通过此校正,可以取消多重检测,方差结果是假阳性率所致。例如,如果 1000 个探针 P <0.05 测验,50 通过了但没有多重检验校正,这只是不超过预计的运气。重要基因名单的实质等同性标准是与 cDNA 芯片实验相同的 [ 5 ] 。
3.13 实时 RT- PCR 验证转录组数据
有两种常用的基因(扩增子)定量检测方法:基因特异荧光探针(如 TaqMan chemistry) 或者特异双链 DNA 结合试剂(SYBR green chemistry ) [22]。我们通过实时 RT-PCR,选择 SYBR green chemistry 来验证 cDNA 芯片发现的 DEG 的表达。不同的基因设计特异引物(见注 32) 。
3.13.1 实时 RT- PCR 反应的准备
( 1 ) PCR 用可视化的 96 孔板在 ABIPRISMA 7500 Sequence Detection System 仪器上进行 ( Applied Biosystems, Foster City, CA,USA ) 。
( 2 ) 总 RNA ( 2 μg 脱氧核糖核酸酶处理过的 RNA,来自 3.4 节)用反转录酶和缓冲液(Superscript EIRT, Invitrogen ) 按照说明手册反转录。
(3 ) PCR 反应用 25 μl 体系:100 ng cDNA,12.5 μl 2X 带 SYBR 绿色荧光染料的 Platinum qPCR Super Mix-UDG ( Invitrogen ) ,0.5 μl ROX 参照染料(Invitrogen ) ,一对特异引物(每个引物 200 ng) ( 见注 33 ) 。
( 4 ) 所有 PCR 反应用了如下标准温度控制:50℃ 2 min , 95°C 2 min , 40 个循环:95℃ 15s 和 60°C 1 min ( 见注 34)。
3.13.2 实时 PCR 数据提取和分析
( 1 ) 原始数据提取。收集每个样品循环阈值(Ct ) [23]。为了比较不同的 cDNA 样本的 Ct 值,所有比较基因的 Ct 值都根据看家基因 actin 进行标准化 (见注释 35) 。
( 2 ) 数据分析。使用 Pfaffl 推荐的方程式计算选择基因的相对表达量 [24] 。这些算法包括校正基因扩展效率。不同目标基因和参照基因的 PCR 扩增效率估计使用 Ramakers 等 [ 25 ] 推荐的方程式。
3.14 递交芯片数据到公共数据库:ArrayExpress
芯片的数据应存放在公共数据库:ArrayExpress [ 26 ] 是在欧洲生物信息研究所( EBI) 的高通量功能基因数据的公共数据库 。这个数据库由两部分组成:ArrayExpress 库(MIAME,主要是初级归档)和 ArrayExpress 数据 库(它是不断注释的选择基因表达谱数据库)。ArrayExpress 是 MGED ( 基因芯片表达数据协会)推荐的三个公共芯片数据库之一。它以保密的形式存储文章使用的数据,允许获得授权的用户,如期刊编辑和审稿入登录数据,文章发表后与其相关的数据可以在指定日期公开。一般情况下,芯片数据提交包括 4 个主要步骤:① 创建一个提交用的新账户(MX 账户);② 提交程序(芯片制备方案、样品的生长和提取步骤、样品标记、杂交,扫描、分析步骤等);③ 提交芯片设计( 名称、设计、技术等) ; ④ 提交实验 ( 实验设计、发表的论文、样品、提取物等)。有关的详细信息请参阅网页: http ://www . ebi. ac. uk/ miamexpress/ Help。
3.15 统计模型
剩余最大似然法(REML 法) [ 27 ] 在 GenStat 第 7 版 ( 2003年)统计系统中应用 ,适合于混合模型( 包括随机和固定效应),以从任何特定比较( 如 14dpa 的 B102-1-1与 L88-31 ) 中,每个基因多达 6 个观察值,建立一套完整的数据集。根据模型偏差估算实验设计中由生物学重复和技术重复所组成的方差,不同的模型之间进行测试时,随自由度变化的其方差变化服从 x2 分布。使用 Wald 检验 [ 28 ] 评估模型中的固定效应,测试统计量也服从 x2 分布。因此,建模考虑了设计上的变异效应( 随机效应)和固定效应 ( 9246 个基因)。经过随机和固定参数(term) 的显著性评估,在 14dpa 的 B 102-1-1 和 L 88-31 比较模型是:
-
投融资
-
精英视角

-
精英视角

-
企业风采

-
会议会展

-
企业风采

-
焦点事件
-
企业风采

-
招标采购

-
焦点事件
-
会议会展

-
科技前沿

-
产品技术

-
科技前沿
-
产品技术
-
科技前沿

-
焦点事件

-
企业风采

-
产品技术

-
焦点事件

-
企业风采

-
产品技术

-
焦点事件

-
焦点事件

-
精英视角

-
综述
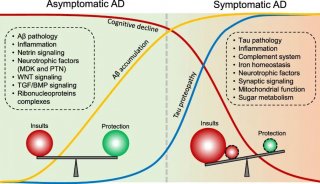
-
会议会展

-
项目成果
-
企业风采

-
焦点事件

-
精英视角

-
产品技术

-
焦点事件

-
科技前沿

-
技术原理
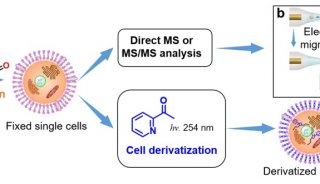
-
项目成果
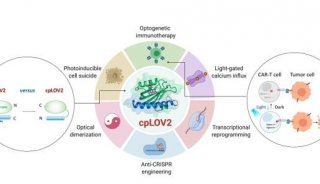
-
会议会展

-
综述
-
焦点事件
-
焦点事件

-
焦点事件
-
会议会展

-
实验室动态

-
会议会展

-
会议会展

-
企业风采
-
焦点事件
-
企业风采

-
焦点事件

-
企业风采

-
焦点事件

-
精英视角

-
企业风采

-
科技前沿

-
焦点事件

-
技术原理

-
企业风采

-
精英视角

-
企业风采

-
企业风采

-
项目成果

-
科技前沿
-
精英视角

-
焦点事件

-
项目成果
-
产品技术

-
焦点事件